マウス系統の遺伝的多様性を解明する
Research Highlight
カテゴリー:遺伝
By Mark Wanner
昨年、完全な「テロメア・トゥ・テロメア」(T2T)ヒトゲノムが完成しました。これにより、今まで「完全」だと考えられていたゲノム配列が、実はまったく完全ではなかったことが強調されることになりました。
ショートリードシーケンシングを超えて
さらに、最近のゲノムの多くは、DNAを通常150~300塩基対の短い断片にするショートリードシーケンス技術を使用して配列決定され、参照配列と比較されます。高速かつ正確で比較的安価ですが、ショートリード手法では通常、ゲノムの大きな部分(全部で約10%)が見落とされます。欠落したセグメントには、GC含量の高い領域のほか、セグメント重複、単純反復、転移因子(TE)などの反復配列が含まれます。TEはゲノム内の他の位置に移動した反復配列であり、これらの配列の可動性はゲノムの変異に大きく寄与します。反復配列は、重複、挿入、欠失、および逆位から生じる構造変異(SV)、すなわちゲノムの差異を形成する基礎となることがよくあります。SVは、ショートリードシーケンスを使用する場合には見落とされることがよくありますが(特に反復によりもたらされるもの)、ゲノム調節異常や疾患において重要な役割を果たす可能性があります。
研究者は、ゲノムをより徹底的に分析するためにロングリードシーケンシングに目を向けています。これらの技術により、はるかに長いDNAセグメントのシーケンシングが可能になり、ゲノムのより完全な全体像を正確に捉えることができるからです。最近の進歩により、ロングリードの精度と有用性が向上し、研究者はヒトの中のゲノムだけでなく、これまで検出されなかったゲノムの特徴を調査できるようになりました。ジャクソン研究所(JAX)とコネチカット大学Health CenterのAssistant Professor Dr. Christine Beck (クリスティーン・ベック)が率いるチームは、もう一つの注目すべき種であるマウスのゲノムを調査し、20種類にわたる多様な近交系の詳細を明らかにしました。これらの系統は、マウスベースの遺伝学およびゲノミクス研究を前進させるうえで不可欠な系統です。
マウス系統間の構造の違い
マウスは、Mus musculus domesticus亜種の系統であるC57BL/6Jの配列に基づいた、GRCm39として知られる独自の参照ゲノムを持っています。しかし、一般的に使用される実験用マウス系統の多くは、他の2つの亜種であるMus musculus castaneusとMus musculus musculusにも由来しており、さまざまな近交系マウスの間には多くの遺伝的差異があります。 Cell Genomicsに掲載された 「Resolution of structural variation in diverse mouse genomes reveals chromatin remodeling due to transposable elements(多様なマウスゲノムの構造変異を解明することにより、転移因子によるクロマチンリモデリングを明らかにする)」で発表された研究において、Dr.ベックは、一般的に使用されている幅広い系統を選択しました。その中には、遺伝的に多様なCollaborative Cross(CC)ならびにDiversity Outbred(DO)マウスパネルのファウンダーマウス7系統、遺伝的起源が不明の異常を有するCCの6系統、異なる遺伝的背景を有する一般的に使用される7系統が含まれていました。
この研究の筆頭著者である大学院生のArdian Ferrajは、これらの20系統のマウスのゲノムを構築し、その配列を使用して、それらのゲノムにC57BL/6Jの参照ゲノムとの違いをもたらす、マウス内に存在するSVを特定しました。Ardianは、ベック研究室のメンバーであるDr. Peter Audanoが開発したプログラムであるPAVを用いて、SVがマウスのゲノム全体に存在し、ゲノム変異に広範囲に寄与していることを示しました。実際、SVには、以前に公開されたさまざまなマウスゲノムの単一ヌクレオチド変異と比較して、影響を受ける塩基数がほぼ5倍含まれています。彼らはまた、ヒトゲノム間よりもマウスゲノム間のSVの多様性がはるかに大きいことも発見し、単一のマウス参照ゲノムではマウス系統全体のゲノムデータをマッピングするには不十分であることを示唆しました。重要なのは、この変異を捕捉するにはロングリードシーケンスが不可欠であるということです。研究チームは、ショートリードのデータと比較してロングリードでは、さらに213,688の挿入、64,277個の欠失、および97の逆位を18のマウス系統にわたって検出しました。
転移因子と構造変異の影響
少数のTEのみがヒトゲノム内で可動性を維持していますが、マウスではより可動性が高くなります。そのため、Dr.ベックと彼女のチームは転移因子バリアント(TEV)に焦点を当て、これがすべてのSVのほぼ40%を占め、その多く(60%)が挿入による変異であることを発見しました。TEVには複数の種類があり、短鎖散在反復配列(SINE)と長鎖散在反復配列(LINE)として知られており、それらは予想されるとおり、そのサイズによって特徴付けられます。マウスゲノムではLINEがSINEの2倍近く多く見られました(47%対24%)。そのサイズのため、LINEはマウスゲノムの可変配列内容のほぼ半分にも寄与していますが、非TEV SVではわずか24%、SINEでは2.1%です。残りの28%のTEVは、さまざまな内在性レトロウイルスの配列によって生成されていました。レトロウイルスは、ゲノムがDNAに逆転写され、その後ゲノムに挿入されるRNAウイルスです。現在のレトロウイルスの多くはエイズやがんなどの疾患に関連していますが、正常な哺乳類のゲノムには、内在性レトロウイルス(ERV)として知られている、数千年にわたるレトロウイルスに由来する大量のDNAが含まれており、これがマウスのゲノム変異を引き起こす要因になっています。
それでは、このすべてのゲノムの変異と活動はどのような結果をもたらす可能性があるのでしょうか? 研究者らは、既知のゲノムの特徴に照らしてSVを調べ、影響の程度を予測しました。遺伝子配列内で新たに検出されたSVのうち、大部分(94,863)はイントロン内にありました。イントロンは、タンパク質の構造を変更しないようにmRNA前駆体から取り除かれた配列です。1,469は遺伝子の両端の非翻訳セグメント(UTR)にあり、510は実際のタンパク質コード配列内にありました。彼らはまた、特定のマウス系統における既知の突然変異サインに関連するDNA修復遺伝子である「Mutyh」という特定の遺伝子内に、これまで検出されていなかったレトロウイルス要素の挿入を見つけました。根本的な変異は不明でしたが、研究チームはこの挿入がMutyh遺伝子発現の大幅な減少と関連していることを発見しました。この発見は、未知のSVが重要なゲノム領域を改変し、健康と機能に関連する形質(疾患を含む)に関連する遺伝子に存在する可能性があることを示しています。
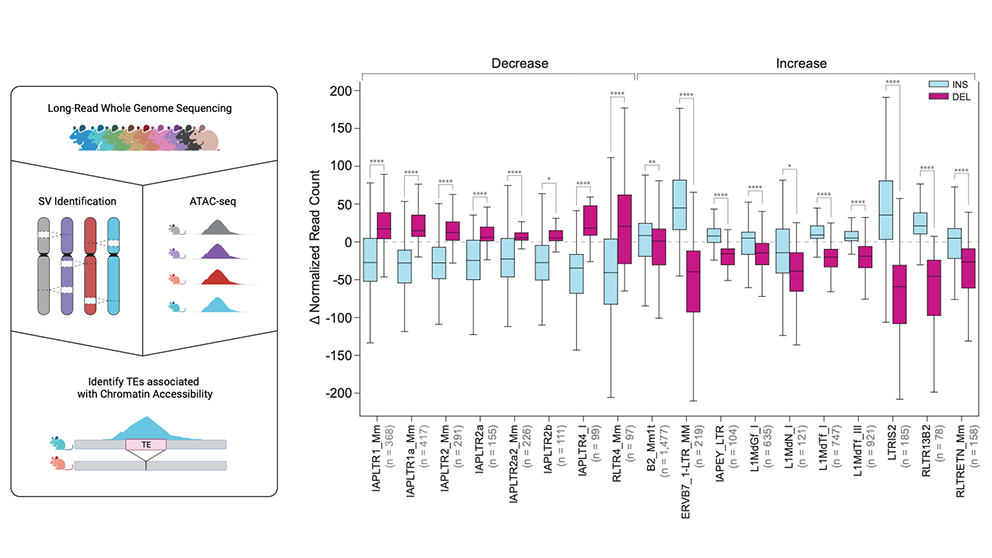
最終的に研究チームは、JAXの研究者であるDr. Laura Reinholdt(ローラ・ラインホルト)と協力して、胚性幹細胞の違いに対するTEの影響を調査しました。TEはゲノムの多様性を促進し、その変異により系統間の遺伝子発現の重要な側面が変化する可能性があります。実際、この研究では、幹細胞クロマチン・アクセシビリティの重大な変化に関連する22,000以上のTEVが発見されました。クロマチン・アクセシビリティは、遺伝的に多様なマウス10系統の胚性幹細胞にわたる、遺伝子発現の重要な調節因子です。研究チームは再び特定の例に焦点を当て、系統に特有のクロマチン・アクセシビリティのシグナルを伴う、遺伝子Slc47a2における系統特異的(CAST/EiJ)イントロン挿入を調査しました。彼らは、イントロン挿入を欠く系統と比較してSlc47a2発現レベルが上昇しており、系統特異的な転写物と多能性因子の結合領域がある可能性を発見し、初期発生におけるTEVの重要な役割を示唆しました。
より完全に理解する
哺乳類の遺伝学とヒトの疾患のモデルとしてのマウスの重要性を考えると、ゲノム変異の機能的影響を十分に理解する必要があります。マウス系統ゲノム間のSVの包括的な検出と特性評価は、理解するうえでの不可欠な部分であり、Dr.ベックとその共同研究者によって生成された結果とデータは、この分野に重要な前進をもたらします。著者らは研究コミュニティ向けに、配列決定されたSVリソース、マウス胚性幹細胞発現リソース、ならびにクロマチン・アクセシビリティのデータを作成しました。これらは、マウスの進化とその特性の基礎となるゲノミクスのさらなる研究に役立つ可能性があります。
著者:
Mark Wanner
米国ジャクソン研究所Research Communications部門Associate DirectorのMark Wannerは、ジャクソン研究所の研究に関するコミュニケーションを統括しています。 サイエンスとコミュニケーション両方のバックグラウンドを持つMark Wannerは、さまざまな媒体で生物医学と臨床科学の問題を取り上げ、それらの情報を多くの視聴者層に発信するとともに、その問題について説明しています。
英語原文: New study reveals details across 20 diverse inbred mouse strains (jax.org)


